Por Peter Dockrill
Publicado na ScienceAlert
Um problema científico de longa data e incrivelmente complexo relativo à estrutura e ao comportamento das proteínas foi resolvido com eficácia por um novo sistema de Inteligência Artificial (IA), relatam os cientistas.
A DeepMind, empresa de Inteligência Artificial sediada no Reino Unido, há anos nos impressiona com seu trabalho de redes neurais em constante avanço que continuamente atacam os humanos em jogos complexos como xadrez e Go.
Todos esses avanços incrementais levaram a muito mais do que dominar diversões recreativas, no entanto.
Em segundo plano, os pesquisadores da DeepMind estavam tentando persuadir suas IAs a resolver enigmas científicos muito mais importantes – como encontrar novas maneiras de combater doenças, prevendo aspectos infinitesimais, mas de vital importância, da biologia humana.
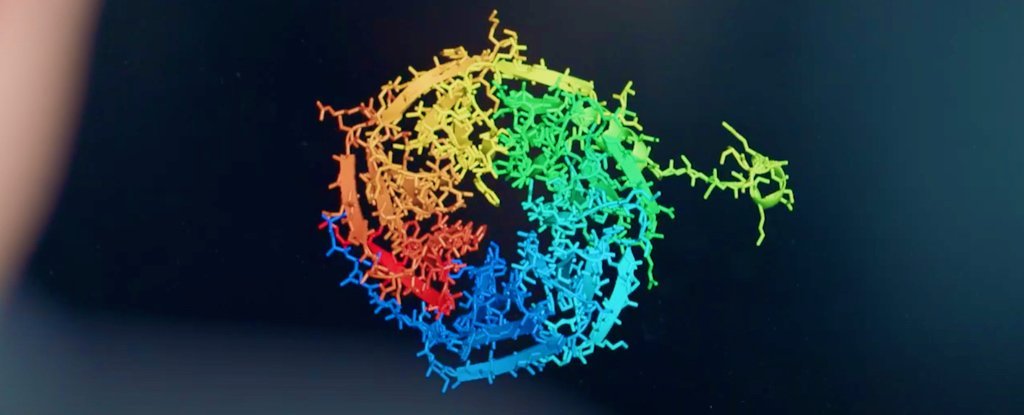
Agora, com a última versão de seu mecanismo AlphaFold AI, eles parecem ter realmente alcançado esse objetivo muito ambicioso – ou pelo menos nos aproximou mais do que os cientistas já fizeram antes.
Por cerca de 50 anos, os pesquisadores se esforçaram para prever como as proteínas alcançam sua estrutura tridimensional, e não é um problema fácil de resolver.
O número astronômico de configurações potenciais é tão assustadoramente grande, na verdade, que os pesquisadores postularam que levaria mais tempo do que a idade do Universo para amostrar todos os arranjos moleculares possíveis.
No entanto, se pudermos resolver esse quebra-cabeça – conhecido como o problema do enovelamento de proteínas – isso constituirá um grande avanço nas capacidades científicas, acelerando enormemente os esforços de pesquisa em coisas como descoberta de medicamentos e modelagem de doenças, e também levando a novas aplicações muito além da saúde.
Por isso, apesar do nível do desafio, há décadas os pesquisadores têm colaborado para avançar no desenvolvimento de soluções para o problema do enovelamento de proteínas.
Um experimento rigoroso chamado CASP (Avaliação Crítica de Técnicas para Predição da Estrutura da Proteína) começou na década de 1990, desafiando os cientistas a desenvolver sistemas capazes de prever os enigmas esotéricos do enovelamento de proteínas.
Agora, em sua terceira década, o experimento CASP parece ter produzido sua solução mais promissora até agora – com o AlphaFold da DeepMind fornecendo previsões de estruturas de proteínas 3D com precisão sem precedentes.
“Estamos presos neste único problema – como as proteínas se enovelam – por quase 50 anos”, disse o cofundador do CASP, John Moult, da Universidade de Maryland (EUA).
“Ver a DeepMind produzir uma solução para isso, tendo trabalhado pessoalmente neste problema por tanto tempo e depois de tantas pausas e começado a se perguntar se algum dia chegaríamos lá, é um momento muito especial”.
No experimento, DeepMind usou uma nova arquitetura de aprendizado profundo para AlphaFold que foi capaz de interpretar e calcular o ‘gráfico espacial’ de proteínas 3D, prevendo a estrutura molecular que sustenta sua configuração enovelada.
O sistema, que foi treinado por meio da análise de um banco de dados de aproximadamente 170.000 estruturas de proteínas, trouxe seu conjunto de habilidades exclusivo para o desafio do CASP deste ano, chamado CASP14, atingindo uma pontuação média em suas previsões de 92,4 GDT (Teste de Distância Global).
Isso está acima do limite de ~90 GDT que geralmente é considerado competitivo com os mesmos resultados obtidos por meio de métodos experimentais, e a DeepMind diz que suas previsões tem uma margem de erro de apenas cerca de 1,6 ångström em média (cerca da largura de um átomo).
“Quase caí da cadeira quando vi esses resultados”, disse o pesquisador de genômica Ewan Birney, do Laboratório Europeu de Biologia Molecular.
“Eu sei como o CASP é rigoroso – ele basicamente garante que a modelagem computacional deve funcionar na tarefa desafiadora de enovelamento de proteínas ab initio. Foi humilhante para nós [humanos] ver que esses modelos poderiam fazer isso com tanta precisão. Haverá muitos aspectos a serem entendidos, mas isso é um grande avanço para a ciência”.
É importante notar que a pesquisa ainda não foi revisada por pares, nem publicada em um jornal científico (embora os pesquisadores da DeepMind digam que isso está a caminho).
Mesmo assim, especialistas que estão familiarizados com a área já estão reconhecendo e aplaudindo o avanço, mesmo que o relatório completo e os resultados detalhados ainda não tenham sido vistos.
“Este trabalho computacional representa um avanço impressionante no problema do enovelamento de proteínas, um grande desafio da biologia de 50 anos”, disse o biólogo estrutural Venki Ramakrishnan, presidente da Royal Society.
“Isso ocorreu décadas antes que muitas pessoas no campo tivessem previsto”.
Os resultados completos ainda não foram publicados, mas você pode ver o resumo da pesquisa, “High Accuracy Protein Structure Prediction Using Deep Learning” (tradução livre: Predição da estrutura da proteína de alta precisão usando aprendizado profundo), aqui e encontrar mais informações sobre o CASP14 aqui.