Quem realmente me conhece sabe que desde que atuava no mercado de trabalho, especificamente na área da informática, sempre estive envolvido com o movimento de Software Livre. Desde os primeiros passos na computação, sempre me interessei sobre o desenvolvimento do GNU/Linux e do movimento Open Source.
Mesmo no início da minha carreira, sempre participei dos projetos de difusão da informática no inicio dos anos 2000, incluindo os movimentos que levaram ao surgimento dos Telecentros em São Paulo. Também promovi palestras e diversos eventos de Install Fest para apresentar o Linux para o público em geral, por sempre me parecer de extrema importância para a difusão tecnológica de forma justa.
Ao decidir migrar da informática clássica para a bioinformática, percebi de imediato que o movimento Open Source era extremamente importante para as práticas científicas e o desenvolvimento de uma Ciência Livre e por isso, direcionei todos os meus esforços no desenvolvimento de uma linha de pesquisa que permitisse a utilização de diferentes ferramentas livres na construção de soluções de bioinformática que possibilitassem a reprodutibilidade dos mais diversos estudos moleculares.
Durante esse período de migração, também percebi um fato importante: as áreas biológicas necessitavam cada vez mais de profissionais com conhecimentos avançados em informática, principalmente programação, e isso ficaria extremamente claro em breve.
Com a chegada do COVID-19, logo percebi que esse era o momento em que minha previsão seria comprovada. Além disso, esse seria o momento em que os programadores (pesquisadores ou não) se encontrariam em uma posição única se comparados com outros pesquisadores clássicos de bancada, onde não apenas poderiam trabalhar normalmente de suas casas durante a pandemia do COVID-19, mas também poderiam ajudar de forma extremamente ativa em diferentes projetos de pesquisa ou mesmo na construção das mais variadas aplicações, com inúmeros objetivos ou graus de importância.
O interessante disso é que esses programadores não precisariam necessariamente ter qualquer conhecimento sobre o COVID-19 ou o SARS-CoV-2, podendo aplicar o seu conhecimento em informática ou estatística em diferentes conjuntos de dados disponíveis livremente e apresentar seus resultados para que os profissionais das áreas biológicas pudessem avaliar se os mesmos fariam sentido.
Assim, uma nova era colaborativa teve inicio e diversos programadores perceberam essa oportunidade.
Coronavírus, Open Source e Open Science
Um caso extremamente curioso pode ser apontado para ilustrar essa minha percepção. Em fevereiro de 2020, um adolescente na área de Seattle foi diagnosticado com COVID-19. Logo depois, os pesquisadores do Seattle Flu Study compartilharam dados genômicos sobre sua cepa do vírus com outros pesquisadores em um site de Ciência Aberta. Armado com esses dados, os pesquisadores envolvidos em um segundo projeto de Ciência Aberta determinaram que a cepa do adolescente era descendente direto de uma cepa do COVID-19 encontrada em um paciente não relacionado na área de Seattle em 20 de janeiro. Essa descoberta foi um elo fundamental para concluir que o vírus estava se espalhando na área de Seattle por semanas.
No caso desse adolescente da área de Seattle, os dados genéticos sobre sua cepa de COVID-19 foram enviados para o Gisaid, uma plataforma para compartilhamento de dados genômicos que busca promover o principio de Open Data. Depois, os pesquisadores da Nextstrain fizeram a conexão com o paciente anterior.
Nextstrain, uma aplicação Open Source
O Nextstrain é um aplicativo de código aberto que rastreia a evolução de vírus e bactérias, incluindo o COVID-19, Ebola, entre outros surtos menos conhecidos, como o Enterovirus D68, usando dados provenientes em grande parte do banco de dados aberto Gisaid. Durante o desenvolvimento do Nextstrain, seus desenvolvedores analisaram os dados compartilhados no Gisaid em busca de mutações e outras informações moleculares/filogenéticas importantes, o que permitiu, por exemplo, identificar a conexão entre os dois casos do COVID-19 em Seattle.
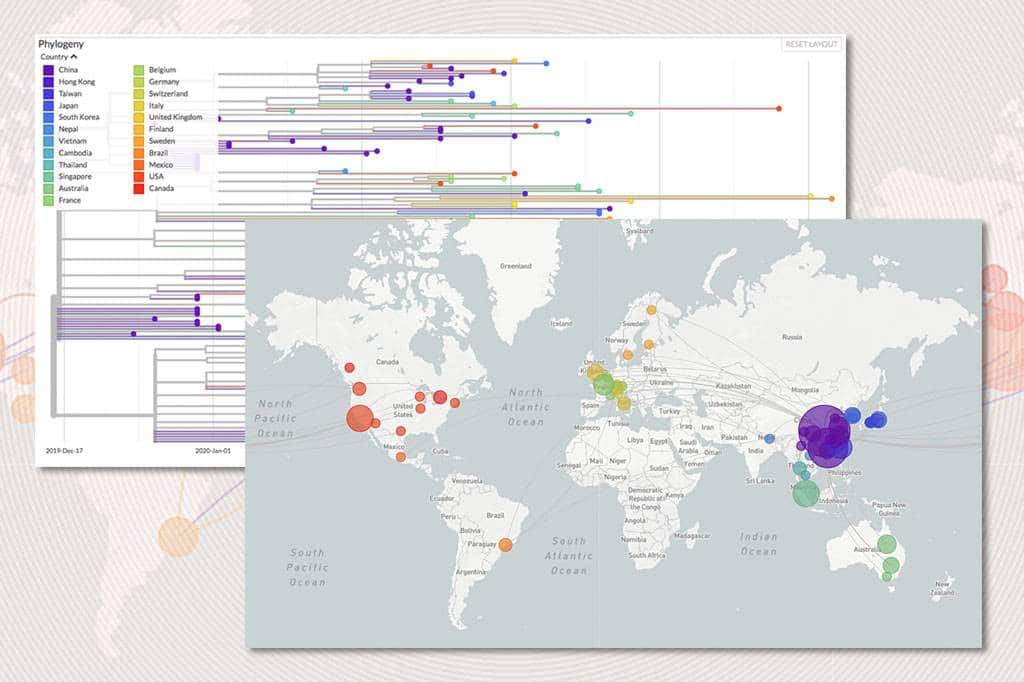
Um ponto importante que deve ser destacado é que o trabalho do Nextstrain só é possível graças ao amplo compartilhamento de dados por cientistas e profissionais de saúde, promovendo assim o conceito de Open Data. Sem esses dados disponíveis, projetos como o Nextstrain não existiriam.
Outro fator que deve ser analisado é que como o código usado pela equipe do Nextstrain é aberto, outros pesquisadores podem criar suas próprias versões do site do Nextstrain ou usar o código como base para novos projetos. Mais importante ainda é que graças a utilização de um código aberto, outros cientistas também podem avaliar a validade científica do trabalho da equipe, permitindo uma revisão aberta e instantânea. Além disso, o próprio código-fonte pode ser atualizado e melhorado por programadores que atuam nas mais diferentes empresas e setores, não restringindo o fazer científico para as Universidades e promovendo o conceito de Ciência Cidadã em um momento de crise mundial.
A metodologia aplicada pela Nextstrain para gerar seus resultados não é nova, sendo apenas a adaptação de metodologias já apresentadas em diferentes trabalhos publicados em periódicos acadêmicos. A grande inovação da equipe do Nextstrain foi perceber que a explosão de dados genômicos disponíveis no Gisaid e a velocidade com que eles são carregados, estaria criando novas oportunidades para preencher a lacuna entre a saúde pública e a Academia, além de permitir que usuários iniciantes também pudessem explorar esses conjuntos de dados, antes restritos ao meio acadêmico.
No entanto, precisamos ter em mente que essas informações e correlações apresentadas pelo Nextstrain não passaram pelo crivo da avaliação e revisão de outros especialistas, o que pode gerar erros de interpretação que podem acabar mais atrapalhando do que ajudando os profissionais de saúde. Esse problema foi evidenciado em uma sequência de postagens do Twitter do cofundador da Nextstrain, que fez correlações sobre duas cepas do COVID-19 baseado simplesmente nas informações de sua aplicação, que depois se mostraram falhas.
Outros projetos Open Source
Existem muitos outros projetos importantes sendo construídos com base no conceito de Open Source e Open Science em resposta ao COVID-19.
Abaixo mais alguns exemplos:
1. CHIME by PennSignals
O Modelo de Impacto Hospitalar COVID-19 para Epidemias (CHIME) é um aplicativo de código aberto criado por cientistas de dados da Penn Medicine da Universidade da Pensilvânia. A ferramenta online permite que os hospitais entendam melhor o impacto que o vírus terá na demanda do hospital.
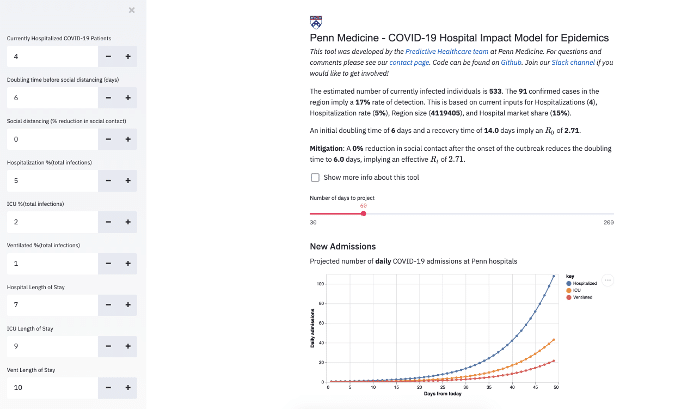
O CHIME pode ser utilizado para obter estimativas de quantos pacientes precisarão de hospitalização, leitos de UTI e ventilação mecânica nos próximos dias e semanas. Um usuário pode inserir, por exemplo, quantos pacientes estão atualmente hospitalizados e ver, com base em outras variáveis, como a demanda pode aumentar nos próximos dias.
O CHIME é desenvolvido em Python e Pandas.
2. Visualizador COVID-19 em tempo real da Locale.ai
Mapas que rastreiam o número de casos nos ajudam a visualizar a escala relativa e a expansão do COVID-19. O Locale.ai criou uma visualização interativa de código aberto de todos os casos conhecidos do COVID-19. O mapa fornece atualizações ao vivo com novos dados à medida que se tornam disponíveis.
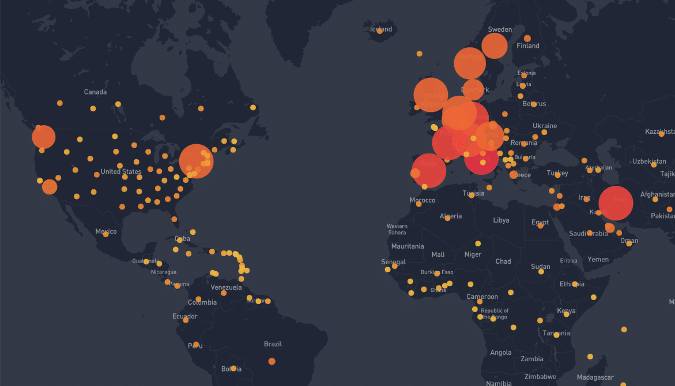
O Locale.ai criou o site de visualização usando o Vue.js , uma estrutura popular que permite que desenvolvedores da Web criem aplicativos modernos. Os dados são recuperados por meio de uma API de código aberto criada pelo usuário do GitHub ExpDev07, que consulta um conjunto de dados de código aberto da Universidade John Hopkins. O conjunto de dados John Hopkins (um agregado de mais de uma dúzia de outras fontes) é atualmente o projeto mais popular relacionado ao COVID-19 no GitHub.
3. DXY-COVID-19-Crawler da BlankerL
O DXY-COVID-19-Crawler foi criado em janeiro e é uma das primeiras respostas da comunidade de código aberto ao COVID-19. Quando o vírus estava se espalhando principalmente na China, a comunidade médica chinesa usava um site chamado DXY.cn para relatar e rastrear casos. Para tornar as informações mais prontamente disponíveis e utilizáveis por outros, o usuário do GitHub BlankerL criou um rastreador da Web para coletar sistematicamente dados do site DXY.cn e disponibilizá-los por meio de uma API e um repositório de dados. Esses dados foram usados por pesquisadores acadêmicos e outros para examinar tendências e visualizar a propagação do vírus.

O DXY-COVID-19-Crawler foi escrito em Python e um pacote chamado Beautiful Soup, que permite aos desenvolvedores extraírem facilmente informações de sites em um processo chamado de web scraping, ou raspagem de dados.
Doe seu poder de computação
Mesmo que você não seja um programador ou profissional de informática, é possível colaborar com um projeto bastante interessante e que auxiliará de uma forma inusitada centenas, senão milhares, de trabalhos e pesquisadores ao redor do mundo.
O Folding @ Home fornece um software, disponível para Windows, macOS e Linux de 64 bits e 32 bits, que é executado em seu computador em segundo plano. Esse software aproveitará o tempo ocioso do seu computador para processar diferentes projetos científicos, incluindo dados do COVID-19, emprestando seus recursos computacionais a esforços científicos para entender melhor esta doença e potencialmente encontrar uma cura.
Fazendo sua parte
Creio que seja inspirador saber que a comunidade de código aberto está respondendo a essa pandemia de uma forma extremamente ativa e aproveitando outras tecnologias de código aberto para trabalhar rapidamente. Sabemos que os próximos dias serão extremamente difíceis e que estamos em uma corrida contra o tempo para achatar a curva do COVID-19, mas os projetos descritos também demonstram que podemos continuar a encontrar motivação na comunidade de código aberto para auxiliar nesse processo.
Por isso, adote um projeto de código aberto, o incentive e colabore quando possível. E caso seja programador, comece a doar um tempo da sua semana para projetos de pesquisa.
Caso tenha interesse, posso indicar diversos projetos ou podemos conversar para o desenvolvimento de um projeto comunitário em diferentes áreas na qual atuo. Sempre existe um espaço para um programador motivado e com vontade de desenvolver!